實務上常常會在分析後看著結果圖,想更改/新增/刪除組別分類看看,
若每次都要重頭跑分析實在太麻煩了,
其實只要獲得品質管制 (Quality Control)得到的三個檔案之後,
調整sample-metadata.tsv後,其程式重繪時間都會大大減低。
本篇結合兩個重點 :
通常重分析廠商可是要多收錢的~
index 與新增一組別 color
sample_name index group color
1St_29_Kenedy_Texas 1St_29_Kenedy_Texas A yellow
1St_152_Metro_North 1St_152_Metro_North A yellow
1St_151_Laramie 1St_151_Laramie B black
1St_141_Mendenhall 1St_141_Mendenhall B brown
本次範例分組與原研究無任何關連,僅示範用途。
reanalyze-group.sh下列是會重繪製會產生的檔案 :
pb-16S-nf 原始無提供各階層熱圖 (Heat map),也藉此繪製產出。
以下步驟會用到 QIIME2-2022.8 程式,若無安裝過可參閱 [Day 06]。
1.建立 reanalyze-group.sh 於 allResults 資料夾下 :
vi reanalyze-group.sh
2.貼上以下資料 :
declare -a hierarchy=("2" "3" "4" "5" "6" "7")
conda activate qiime2-2022.8
mkdir group_results
qiime taxa barplot \
--i-table dada2/dada2-ccs_table_filtered.qza \
--i-taxonomy results/best_tax.qza \
--m-metadata-file ../sample-metadata.tsv \
--o-visualization group_results/taxa-bar-plots.qzv
#物種分配 (Taxonomy assignment) 後的分類柱狀圖
for ((i=0;i<${#hierarchy[@]};i++))
do
qiime taxa collapse \
--i-table dada2/dada2-ccs_table_filtered.qza \
--i-taxonomy results/best_tax.qza \
--p-level ${hierarchy[$i]} \
--o-collapsed-table group_results/col_table_${hierarchy[$i]}.qza
qiime feature-table heatmap \
--i-table group_results/col_table_${hierarchy[$i]}.qza \
--o-visualization group_results/heatmap_${hierarchy[$i]}.qzv \
--p-color-scheme YlOrRd \
--p-cluster features
echo "Level ${hierarchy[$i]} heatmap is done."
done
#熱圖 (Heat map)
qiime diversity core-metrics-phylogenetic \
--i-phylogeny results/phylogeny_diversity/phylotree_mafft_rooted.qza \
--i-table dada2/dada2-ccs_table_filtered.qza \
--p-sampling-depth 32346 \
--m-metadata-file ../sample-metadata.tsv \
--output-dir group_results/core-metrics-results
echo "core-metrics-phylogenetic (alpha and beta Diversity) is done."
#組內物種多樣性 (Alpha diversity)與組間物種多樣性 (Beta diversity)
#--p-sampling-depth 為取樣深度
qiime diversity alpha-rarefaction \
--i-table dada2/dada2-ccs_table_filtered.qza \
--i-phylogeny results/phylogeny_diversity/phylotree_mafft_rooted.qza \
--p-max-depth 48233 \
--m-metadata-file ../sample-metadata.tsv \
--o-visualization group_results/alpha-rarefaction.qzv
echo "Alpha-rarefaction is done."
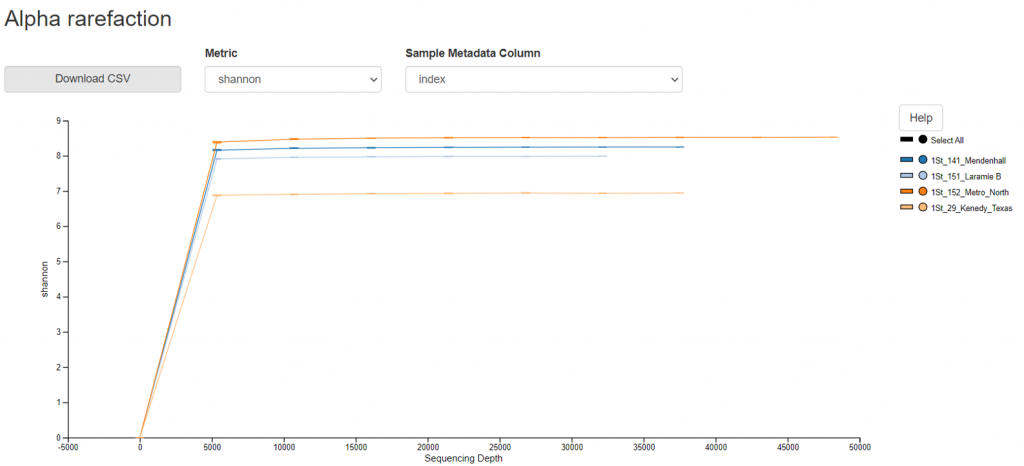
#組內物種多樣性 (Alpha diversity)的稀疏曲線(rarefaction)
#--p-max-depth 為所有樣本中條數最大的值
要特別注意的是取樣深度與最大深度會隨著不同樣本而異需要調整,
可至 [Day 15] 有詳細解說。
此外熱圖就不輸出 Level 1 (界 Kingdom) 的部份了,
想想這階層的熱圖其實蠻冗的 XD
reanalyze-group.sh. reanalyze-group.sh
完成的樣子 :
因為圖實在太多了,真是奢侈的煩惱,就不全部秀出,主要提供分析者檢視相關路徑 :
物種分配 (Taxonomy assignment) 後的分類柱狀圖TGS_sewage/allResults/group_results/taxa-bar-plots.qzvTGS_sewage/allResults/results/taxa_barplot.qzv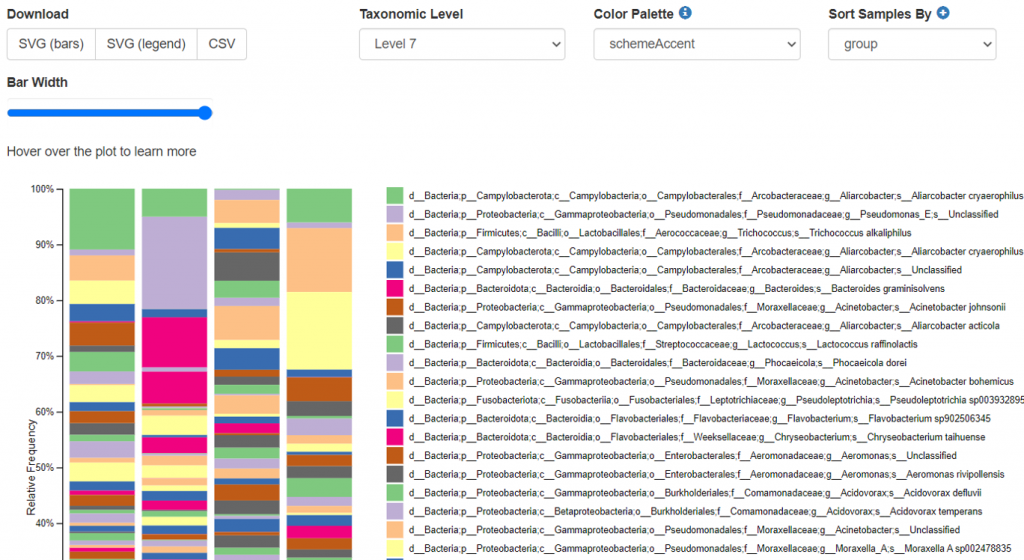
熱圖 (Heat map)TGS_sewage/allResults/group_results/heatmap_5.qzv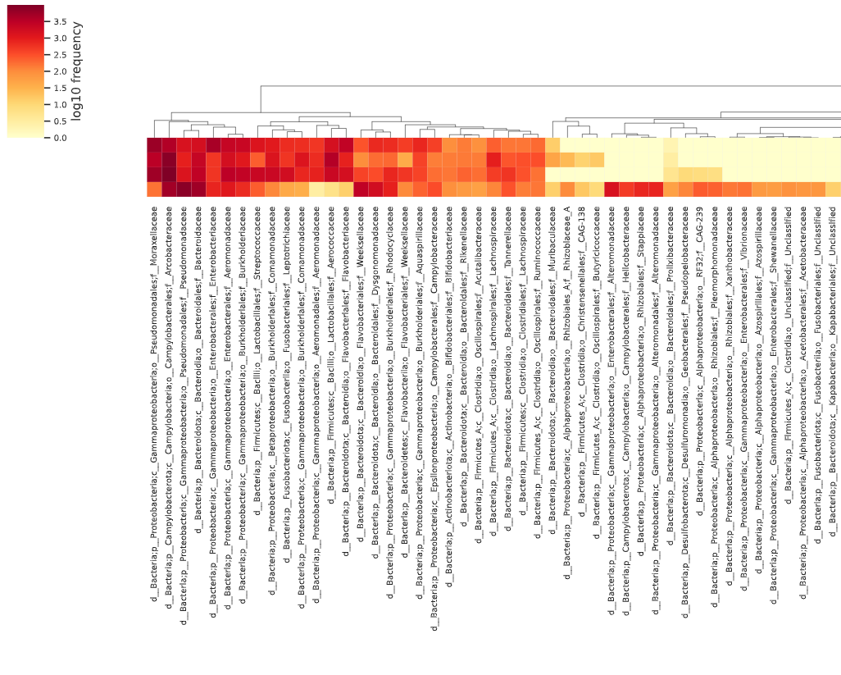
組內物種多樣性 (Alpha diversity) - 稀疏曲線TGS_sewage/allResults/group_results/alpha-rarefaction.qzv
TGS_sewage/allResults/results/alpha-rarefaction-curves.qzv
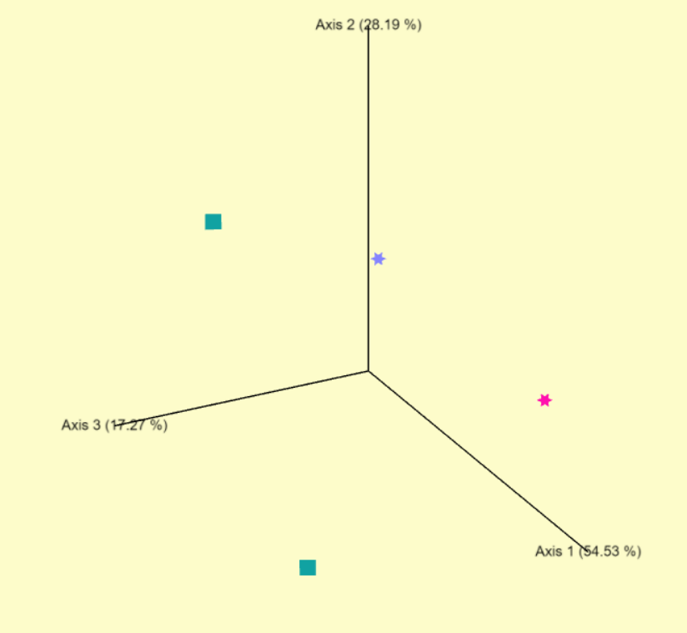
組間物種多樣性 (Beta diversity)TGS_sewage/allResults/group_results/core-metrics-diversity/
TGS_sewage/allResults/results/phylogeny_diversity/core-metrics-diversity/